
« Systems Biology Core Facility »
La compréhension, à l’échelle moléculaire, de la pathogénèse de maladies précédemment étiquetées « idiopathiques » a grandement bénéficié du développement récent de techniques puissantes d’analyses « -omiques ». En particulier, la caractérisation du génome humain et le perfectionnement de techniques de séquençage ont permis l’identification de mutations à l’origine de nombre de maladies monogéniques et le développement plus récent de scores de risque polygéniques pour les maladies courantes. Cependant, on sait que les variations à l’échelon du génome, prises isolément, ne sont que peu corrélées au phénotype clinique, en raison de multiples régulations au niveau et en aval de l’expression du gène. Tenir compte de cette régulation complexe (épigénétique, post-transcriptionnelle et -traductionnelle) nécessite le recours à des strates supplémentaires d’analyses, entre autres au niveau du transcriptome, protéome et métabolome. L’intégration de ces données « multi-omiques » avec le phénotype clinique, voire des données environnementales (l’« exposome ») permet une approche holistique, non-biaisée de la compréhension de maladies complexes (1). Elle pose cependant le défi de l’analyse de jeux de données qui nécessitent le développement d’outils bioinformatiques sophistiqués, avec l’appoint éventuel d’algorithmes d’intelligence artificielle (2).
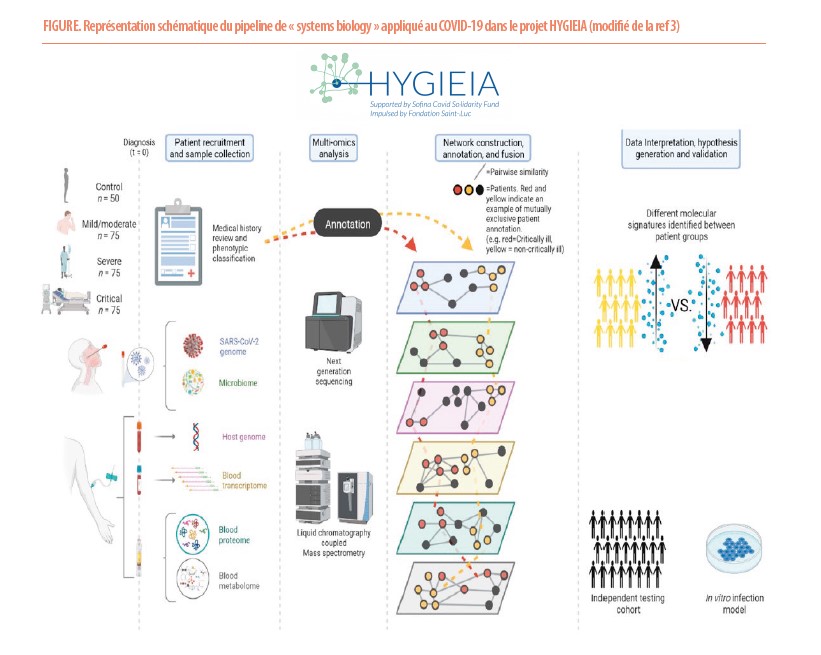
Pour relever ce défi, plusieurs acteurs du Secteur des Sciences de la Santé (SSS/UCLouvain et Cliniques universitaires Saint-Luc-campus de Woluwé) ont décidé de mettre en commun leurs expertises pour constituer une filière unique, allant du malade à l’identification de « nœuds » physiopathologiques de sa maladie par une approche de « Systems Biology », ou biologie des systèmes. Cette filière (ou « pipeline ») comprend la collecte de données cliniques et d’échantillons biologiques des patients, le traitement pré-analytique de ces échantillons, l’analyse de ceux-ci par les techniques les plus récentes de génomique, méta-génomique, transcriptomique, protéomique et métabolomique, l’intégration des résultats dans une banque de données multi-modale et leur traitement bioinformatique utilisant, entre autres, une analyse des « réseaux d’interaction » (« network biology » ; 1,2), inspirée des théories mathématiques d’analyse des graphes complexes.
Monté initialement grâce à un financement du « Sofina Solidarity Fund » via la Fondation Saint-Luc, qui a permis de renforcer l’équipement et le personnel des plateformes « omiques » existantes, ce « pipeline » a été (et est encore) appliqué à l’étude de la physiopathologie de l’infection au SARS-COV-2, et en particulier aux mécanismes sous-tendant les formes critiques du COVID ainsi que le développement du syndrome de « COVID long » (projet HYGIEIA (3). Initialement monocentrique, le recrutement pour cette étude a été étendu au Grand Hôpital de Charleroi (GHDC), CHU Namur-Mont-Godinne, Clinique de l’Europe et Clinique Saint-Pierre, Ottignies. Plus largement, le projet est intégré dans le portefeuille de projets « MedReSyst » (« Medecine des Réseaux et des Systèmes » ; https://medresyst.org/) de l’Initiative d’Innovation Stratégique de la Région Wallonne dans le cadre des financements FEDER de la Commission Européenne.
Forts de cette expansion, les fondateurs du projet ont décidé d’ouvrir l’accès au « pipeline » aux cliniciens et chercheurs académiques qui souhaitent appliquer ce paradigme à leur recherche clinique ou fondamentale, en créant la « Systems Biology Core Facility » au sein du Secteur. Cette nouvelle plateforme est « trans-institutionnelle », puisqu’elle implique des personnes et structures relevant de l’IREC, LDRI et DDUV, ainsi que des Cliniques Saint-Luc (voir ci-dessous).
Les modalités d’accès se veulent simples et flexibles : le responsable académique intéressé remplit un formulaire interactif disponible sur le site Web de la plateforme SYSBIOL(https://uclouvain.be/sysbiol) qui sert de guichet unique ; un « steering committee » évalue le projet et organise au besoin une réunion avec le demandeur pour préciser les objectifs et modalités ; en fonction, un devis est calculé et un calendrier fixé.
La plateforme fonctionne suivant un schéma « fee-for-service » et propose des prix tenant compte des développements méthodologiques et des coûts réels qui se veulent avantageux par rapport au « benchmark » (e.a. commercial), tout en garantissant la couverture des frais de fonctionnement et de personnel au prorata de l’utilisation. L’utilisateur peut opter pour des analyses mono- ou multi-omiques, avec ou sans analyse bioinformatique/biostatistique en aval.
Partenaires de la « Systems Biology Core Facility »
Profs J-C Yombi, L. Belkhir, Drs J. De Greef, L. Bamps, Service de Médecine Interne et maladies infectieuses, Cliniques universitaires Saint-Luc et Institut de Recherche Expérimentale et Clinique (IREC), UCLouvain
Profs. L. Elens (PGMK), Prof. P. Cani (MNUT), Bradley Ward, Louvain Drug Research Institute (LDRI), Dr. S. Pyr dit Ruys, Institut de Recherche Expérimentale et Clinique (IREC), UCLouvain
Profs. B. Kabamba (MBLG), V. Haufroid (LTAP), J. Dewulf (LTAP Cliniques universitaires Saint-Luc et Institut de Recherche Expérimentale et Clinique (IREC), UCLouvain
Prof. G. Bommer, Dr. D. Vertommen (MASSPROT), Institut de Duve, UCLouvain
Prof. L. Gatto (CBIO), Institut de Duve, UCLouvain,
Prof. S. Jodogne (INGI/ICTM/SST), UCLouvain
Références
- Silverman EK, Schmidt HHHW, Anastasiadou E, Altucci L, Angelini M, Badimon L, Balligand JL, et al. Molecular networks in Network Medicine: Development and applications. Wiley Interdiscip Rev Syst Biol Med. 2020 Nov;12(6):e1489. doi: 10.1002/wsbm.1489
- Maron BA, Altucci L, Balligand JL, Baumbach J, Ferdinandy P, Filetti S, et al. ; International Network Medicine Consortium. A global network for network medicine. NPJ Syst Biol App. 2020 Aug 31;6(1):29. doi: 10.1038/s41540-020-00143-9.
- Ward B, Yombi JC, Balligand JL, Cani PD, Collet JF, de Greef J, et al. HYGIEIA: HYpothesizing the Genesis of Infectious Diseases and Epidemics through an Integrated Systems Biology Approach. Viruses. 2022 Jun 23;14(7):1373. doi: 10.3390/v14071373.
Affiliations
Pole de Pharmacologie et Thérapeutique (FATH), Institut de Recherche Expérimentale et Clinique (IREC), UCLouvain
Correspondance
Pr Jean-Luc Balligand Cliniques universitaires Saint-Luc Service de Médecine Interne et maladies infectieuses Avenue Hippocrate 10 B-1200 Bruxelles