L’intelligence artificielle accélère le développement de nouveaux traitements pour les maladies rares
Olivier Devuyst
L’intelligence artificielle (IA) joue un rôle de plus en plus important dans la découverte de médicaments. Les progrès dans la génération de modèles génétiques et dans l’utilisation de larges datasets et des algorithmes d’apprentissage permettent de mieux comprendre certaines maladies rares et de développer plus rapidement de nouvelles approches thérapeutiques (1). Le groupe dirigé par le Pr. Olivier Devuyst a ainsi démontré, dans une étude publiée dans ’Nature Communications’, l’utilité de l’IA pour cibler les causes et les traitements possibles d’une maladie rare dévastatrice, la cystinose (Figure 1) (2).
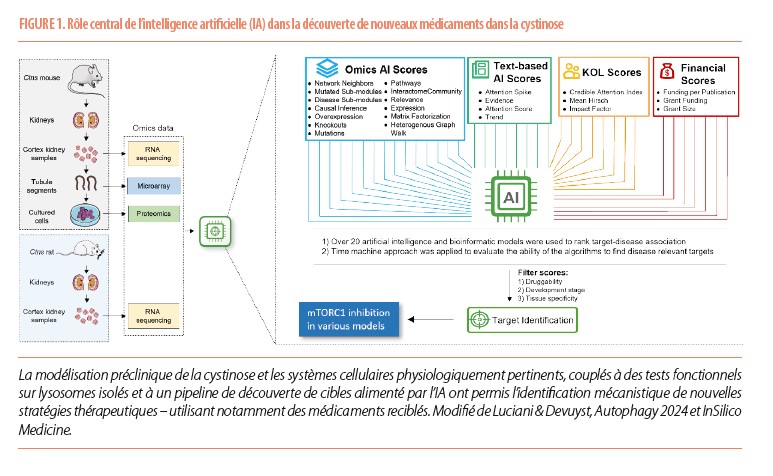
La cystinose est une maladie lyosomale rare qui touche environ 1 nouveau-né sur 100.000 à 200.000 dans le monde. La maladie, transmise en mode récessif, est causée par la perte de fonction de la cystinosine, qui exporte normalement la cystine hors des lysosomes. Il s’agit donc d’une forme typique de maladie de surcharge des lysosomes. La cystinose néphropathique, la forme la plus courante et la plus grave de la maladie, se manifeste par une dysfonction tubulaire des reins au cours des premiers mois de la vie, conduisant à de multiples complications métaboliques et le développement rapide d’une insuffisance rénale avant l’âge de 10 ans. Les enfants atteints de cystinose souffrent de complications multisystémiques, sans traitement curatif à ce jour. Le traitement par la cystéamine, qui vise à dépléter le contenu lysosomal en cystine, est très lourd, grevé d’effets secondaires et d’une faible compliance, sans effet sur certaines complications majeures de la maladie – dont l’atteinte tubulaire.
Le travail, mené en collaboration avec Insilico Medicine, une société qui utilise l’IA pour la découverte de médicaments, visait à découvrir le mécanisme cellulaire sous-jacent à l’atteinte tubulaire rénale de la cystinose. En s’appuyant sur des organismes modèles (rat, souris, zebrafish) et sur la plateforme PandaOmics d’Insilico, les voies pathogènes et les cibles thérapeutiques prioritaires dans les cellules de la cystinose ont été identifiées. En particulier, les résultats ont révélé que le stockage de la cystine dans les lysosomes stimule l’activation de la protéine mTORC1, ce qui entraîne une altération de la différenciation et de la fonction des cellules tubulaires rénales (2,3).
En utilisant la plateforme PandaOmics, l’équipe a pu produire une liste de médicaments existants qui pourraient être réaffectés au traitement de la cystinose à court terme. Pour ce faire, elle a analysé la structure des médicaments, les enzymes cibles, les effets secondaires potentiels et l’efficacité dans les tissus affectés. La rapamycine, médicament déjà autorisé, a ainsi été identifiée comme un candidat prometteur pour le traitement de la cystinose. Des études sur des systèmes cellulaires et des organismes modèles ont confirmé que le traitement à la rapamycine restaurait l’activité des lysosomes et rétablissait les fonctions cellulaires (Figure 1).
Ces résultats démontrent le potentiel de l’IA, combinée à des systèmes biologiques et des logiciels d’apprentissage dans la découverte de nouveaux traitements, parfois basés sur des molécules connues de longue date. Ces méthodes pourraient s’avérer décisives dans le domaine des maladies génétiques rares, dont plus de 90% restent sans traitement.
Références
- Pun FW, Ozerov IV, Zhavoronkov A. AI-powered therapeutic target discovery. Trends Pharmacol Sci. 2023 Sep;44(9):561-572.
- Berquez M, Chen Z, Festa BP, Krohn P, Keller SA, Parolo S, Korzinkin M, Gaponova A, Laczko E, Domenici E, Devuyst O, Luciani A. Lysosomal cystine export regulates mTORC1 signaling to guide kidney epithelial cell fate specialization. Nat Commun. 2023 Jul 14;14(1):3994.
- Luciani A, Devuyst O. The CTNS-MTORC1 axis couples lysosomal cystine to epithelial cell fate decisions and is a targetable pathway in cystinosis. Autophagy. 2024 Jan;20(1):202-204.
Recherche clinique à partir des dossiers patients informatisés : outils innovants aux CuSL dans le respect de la protection des données sensibles
Joëlle Thonnard, Aline van Maanen, Audrey Timmermans, Yannick Barussaud, Cédric Van Marcke, Olivier Devuyst, Demet Yuksel, Marianne Philippe
La digitalisation des données de santé acquises dans les hôpitaux et chez les médecins constitue une source de données en vie réelle (RWD – Real World Data), générant un potentiel énorme pour répondre à des questions de santé publique (RWE – Real World Evidence). Cependant, la diversité des formats de données et les règlements concernant la protection des données à caractère personnel compliquent la mise en route des recherches. Des projets sont lancés pour développer et déployer des solutions à ces obstacles tant au niveau national, européen et international (1). Les cliniques universitaires Saint-Luc (CuSL) s’inscrivent dans cette démarche avec deux approches complémentaires par la participation au projet Digione et Data Capabilites.
L’Institut Roi Albert II (IRA2), le Département d’Information Médicale (DIM), le Département d’Information et Système (DIS) et l’Unité de Support Statistique (USS) collaborent dans le projet Digione avec cinq autres hôpitaux européens. Ce projet est financé par le consortium DIGICORE, un organisme focalisé sur la recherche en oncologie (2). Une plateforme sécurisée est déployée pour accueillir un entrepôt de données (EDS ou Data Warehouse) au format commun OMOP-CDM (Observational Medical Outcomes Partnership - Common Data Model) (3). Dans ce type de projet, des données du Dossier Patient Informatisé (DPI) sont extraites et transformées pour peupler l’EDS. Les données dé-identifiées mises au format OMOP sont consultables par les chercheurs autorisés afin d’effectuer des évaluations de cohortes. Les données peuvent de plus être exploitées dans le cadre d’études cliniques approuvées par les Comités d’Ethiques. L’analyse des données repose sur une approche fédérée, c’est-à-dire que les données individuelles restent dans chaque l’hôpital, le chercheur envoie son algorithme d’analyse aux hôpitaux participants, lesquels partagent leurs résultats statistiques respectifs avec le chercheur qui réalise ensuite une méta-analyse des résultats des différents hôpitaux. Selon le même principe, un chercheur peut envoyer des algorithmes basés sur l’intelligence artificielle (IA) (4) (Figure 1).

L’Institut des Maladies Rares (IMR), le DIM, le DIS et l’USS collaborent aussi dans le projet SPECTRE-HD avec l’UZ Brussel, UZ Antwerpen et Ziekenhuis Oost-Limburg. Ce projet est financé par le SPF santé publique dans le cadre des projets Data Capabilities (5). Dans ce projet, deux plateformes sécurisées formant un « Trusted Research Environment » (TRE) sont développées : une interne à chaque hôpital et une externe partagée entre les hôpitaux. Ce type de plateforme sécurisée fournit un accès contrôlé à des données sensibles dé-identifiées (dans des formats divers, voire textuels), et des outils analytiques, statistiques et d’IA. Les chercheurs ont accès uniquement aux données pour lesquels ils ont demandé et obtenu l’autorisation. Ils peuvent exporter les résultats (et donc les données agrégées), mais pas les données individuelles. Les accès et les flux de données sont tracés et contrôlés. La plateforme fonctionne dans le cadre des 5S : « Safe data, Safe projects, Safe people, Safe settings, Safe outputs » (6). Dans le futur, ce type de plateforme pourrait être accessible pour des analyses fédérées (7) (Figure 2).
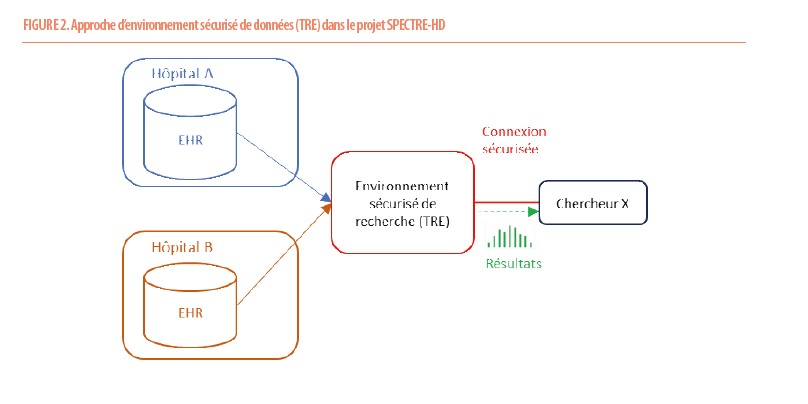
Les deux projets en cours serviront de rampe de lancement pour des recherches cliniques et/ou basées sur l’IA dans d’autres domaines diagnostiques et thérapeutiques. Ils démontreront également à tous nos collaborateurs l’intérêt et l’importance de tenir les dossiers cliniques rigoureusement à jour, et d’éviter autant que possiblement les données dictées ou écrites uniquement sous forme de texte libre. Les CuSL se placent donc en bonne position pour participer à des projets collaboratifs de recherche exploitant les données du monde réel à partir des dossiers patients informatisés. Ceci se fera toujours dans le respect strict de ce que les patients consentent lors de leur prise en charge en routine clinique. En effet, à moins d’un refus clairement notifié, la prise en charge dans notre hôpital universitaire implique que leurs données dé-identifiées puissent être utilisées pour remplir des missions d’enseignement clinique et de recherche scientifique.
Références
- Plan d’action e-santé 2022-2024 actieplan_egezondheid_2022-2024_protocolakkoord.pdf (belgium.be)
- Consortium DIGICORE DigiCore Projects (digicore-cancer.eu)
- OMOP-CDM https://www.ohdsi.org/data-standardization/
- Cremonesi F, Planat V, Kalokyri V, Kondylakis H, Sanavia T, Miguel Mateos Resinas V, Singh B, Uribe S. The need for multimodal health data modeling: A practical approach for a federated-learning healthcare platform. J Biomed Inform. 2023 May;141:104338.
- Projets Data Capabilities Appel à projets « Data Capabilities » | SPF Santé publique (belgium.be)
- Trusted Research Environment Trusted Research Environment service for England - NHS Digital
- Multi-party trusted research environment federation: Establishing infrastructure for secure analysis across different clinical-genomic datasets - DARE UK
Affiliations
Cliniques universitaires Saint-Luc, B-1200 Bruxelles
1. Institut des Maladies Rares
2. Département d’Information Médicale
3. Unité de Support Statistique
4. Département d’Information et Système
5. Institut Roi Albert II (IRA II)
Correspondance
Pr Olivier Devuyst
Institut des Maladies rares
Cliniques universitaires Saint-Luc
Avenue Hippocrate 10
B-1200 Bruxelles